De nouveaux antibiotiques actifs sur le staphylocoque doré résistant à la méticilline
Le Staphylococcus aureus résistant à la méticilline (SARM, en anglais MSRA) est un staphylocoque doré qui résiste à de nombreuses classes d’antibiotiques (comme les puissantes β-lactamines) utilisables contre le Staphylococcus aureus ordinaire, non résistant. Les infections dues au SARM posent de graves problèmes de santé publique. Aussi de nouvelles stratégies s’avèrent nécessaires. Une équipe de chimistes et de biologistes de l’Université Notre Dame à Indiana, USA vient de découvrir une nouvelle classe d’antibiotiques différente de celle des β-lactamines, les oxadiazoles, qui sont actifs contre le SARM et d’autres bactéries Gram- positives.
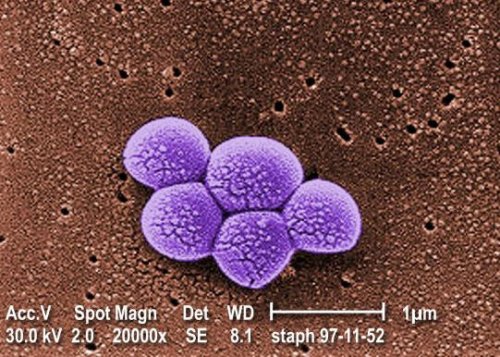
Fig.1.Micrographie colorée obtenue au microscope électronique à balayage de la bactérie Staphylococcus aureus SARM. Grossissement 20000 fois.
Crédit CDC. Janice Carr.
Les staphylocoques, bactéries très courantes, ne causent généralement pas d’infections sur les personnes en bonne santé. Ils peuvent néanmoins entraîner des infections de la peau et des plaies, ou des pneumonies et des infections du sang (septicémie). Le SARM n’est pas plus virulent dans ses effets que les autres staphylocoques, mais son traitement est bien plus problématique. En effet, jusqu’ici, les seuls produits efficaces contre le SRAM étaient au nombre de trois, dont un seul, le linelozide, pouvait être administré oralement. Et une résistance à ceux-ci a déjà émergé.
La résistance aux β-lactamines du SARM provient de la présence dans sa paroi cellulaire d’une « protéine de liaison aux pénicillines », PLP, particulière, la PLP2a. Le Staphylococcus aureus non résistant a dans sa membrane cellulaire quatre plP qui sont, elles, inhibées par la méticilline. La paroi cellulaire alors affaiblie éclate et entraîne la mort de la cellule. Il n‘en est pas de même avec le SARM, où la PLP2a résiste à l’attaque par tous les antibiotiques de la classe des β-lactamines, comme la méticilline.
La liaison peptidique est le résultat de la réaction entre la fonction acide carboxylique COOH d’un acide aminé avec la fonction amine NH2 d’un autre acide aminé.
Les chercheurs se sont fixés pour but de trouver un antibiotique qui inhiberait la protéine PLP2a du SARM. Pour cela, ils ont d’abord pratiqué une sélection in silico, c’est-à-dire à l’ordinateur de 1,2 millions de composés éventuellement inhibiteurs de PLP2a.
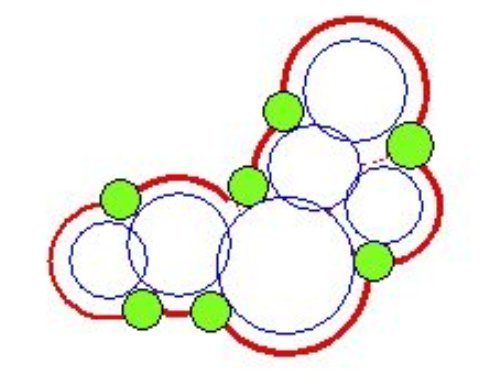 La surface de la molécule est l’enveloppe des boules de Van der Waals cerclées en bleu. La « surface accessible au solvant » (en rouge) correspond à l’ensemble des positions possibles du centre d’une boule de solvant (en vert) qui roulerait sur la surface de Van der Waals de la molécule. C’est à l’aide de cette surface que l’on calcule les interactions avec une autre molécule.
La surface de la molécule est l’enveloppe des boules de Van der Waals cerclées en bleu. La « surface accessible au solvant » (en rouge) correspond à l’ensemble des positions possibles du centre d’une boule de solvant (en vert) qui roulerait sur la surface de Van der Waals de la molécule. C’est à l’aide de cette surface que l’on calcule les interactions avec une autre molécule.
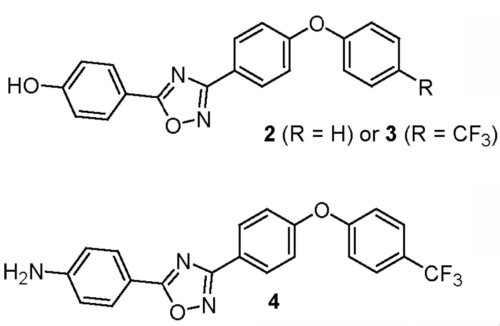
La structure de chacun des composés était combinée avec celle obtenue aux rayons-X de la protéine PLP2a. Un programme classe les composés selon leur plus forte interaction avec PLP2a. La sélection résultante fournit 29 composés. Ils furent testés pour leur activité antibactérienne envers une série de bactéries. Le composé 1, un oxadiazole, dont on voit la structure chimique en bas à droite de la figure 2 présentait une activité contre S. aureus et Enterococcus faecium. On a alors testé 370 variantes de ce composé 1.
La figure 2 ci-dessous montre la structure de la protéine PLP2a de SARM (en vert) sous la forme de ce que les chimistes appellent la surface accessible au solvant. (voir encadré précédent) et l’interaction avec ce composé.
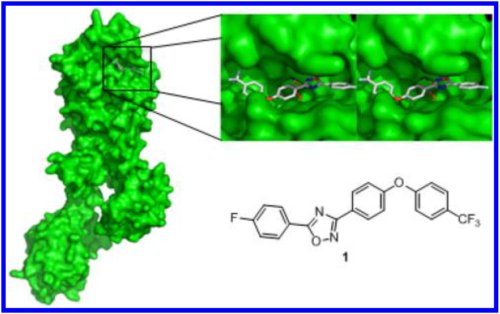
Fig. 2. La structure obtenue aux rayons-X (à gauche) est représentée
sous la forme d’une surface accessible au solvant. En haut à droite,
on a montré en perspective son interaction avec le composé 1, un oxadiazole,
(en bas à droite) prédit par la sélection informatique. Crédit JACS.
.On a de nouveau sélectionné 3 de ces composés qui, après des tests in vitro et in vivo, s’avérèrent capables d’inhiber la protéine PLP2a du SARM, affaiblissant ainsi la paroi cellulaire de la bactérie. Ces trois oxadiazoles ont la structure représentée ci-dessous :
On a testé in vivo sur des souris les propriétés de ces trois oxadiazoles, les composés 3 et 4 ont de meilleures propriétés pharmacodynamiques et leur administration orale est efficace à 100%. Ils ont tous les deux des effets sur le SARM et le composé 3 a une efficacité supérieure pour le même dosage.
Comme le font remarquer les professeurs Maylang Chang et S. Mobashery, c’est grâce à un très long travail préparatoire sur le problème de la résistance du SARM qu’a pu être mise au point la stratégie qui a conduit à l’obtention de ces nouveaux antibiotiques de la classe des oxadiazoles.
Pour en savoir plus :
Discovery of a New Class of Non-β-lactam Inhibitors of Penicillin-Binding Proteins with Gram-Positive Antibacterial Activity
Peter I. O’Daniel, Zhihong Peng, Hualiang Pi, Sebastian A. Testero, Derong Ding, Edward Spink,
Erika Leemans, Marc A. Boudreau, Takao Yamaguchi, Valerie A. Schroeder, William R. Wolter,
Leticia I. Llarrull, Wei Song, Elena Lastochkin, Malika Kumarasiri, Nuno T. Antunes,
Mana Espahbodi, Katerina Lichtenwalter, Mark A. Suckow, Sergei Vakulenko, Shahriar Mobashery,
and Mayland Chang
J. Am. Chem. Soc. 2014, 136, 3664−3672